4 个版本
| 0.2.2 | 2024年3月27日 |
|---|---|
| 0.2.1 | 2024年2月27日 |
| 0.2.0 | 2024年2月20日 |
| 0.1.0 | 2024年2月6日 |
在 科学 类别中排名第 994
每月下载量 280
150KB
503 行代码(不包括注释)
gxf2bed
周围最快的 G{F,T}F 到 BED 转换工具!
翻译
chr27 gxf2bed gene 17266470 17285418 . + . gene_id "ENSG00000151743";
chr27 gxf2bed transcript 17266470 17281218 . + . gene_id "ENSG00000151743"; transcript_id "ENST00000541931.8";
chr27 gxf2bed exon 17266470 17266572 . + . gene_id "ENSG00000151743"; transcript_id "ENST00000541931.8"; exon_number "1"; exon_id "ENST00000541931.8.1";
...
成
chr27 17266469 17281218 ENST00000541931.8 1000 + 17266469 17281218 0,0,200 2 103,74, 0,14675,
在你眨眼之间!
转换
- 智人 GRCh38 GENCODE 44 (252,835 个转录本) 需要 2.99 秒。
- 小鼠 GRCm39 GENCODE 44 (149,547 个转录本) 需要 1.91 秒。
- 家犬 ROS_Cfam_1.0 Ensembl 110 (55,335 个转录本) 需要 0.95 秒。
- 鸡 bGalGal1 Ensembl 110 (72,689 个转录本) 需要 1.07 秒。
版本 0.2.2 的新功能
- 允许在不具有层次关系的情况下进行翻译,以与 Prokka 输出兼容
- 要执行平实的转换而不使用层次结构,只需显式声明
--parent """" --child """"
用法
Usage: gxf2bed[EXE] --input/-i <GTF/GFF> --output/-o <BED> [--parent/-p <PARENT>] [--child/-c <CHILD>] [--feature/-f <FEATURE>]
Arguments:
--input/-i <GTF/GFF>: a .gtf/.gff file
--output/-o <BED>: path to output .bed file
--parent/-p <PARENT>: parent node [default: "transcript"]
--child/-c <CHILD>: child node [default: "exon"]
--feature/-f <FEATURE>: feature to extract from the attribute line [default: "transcript_id"]
Options:
--help: print help
--version: print version
--threads/-t: number of threads (default: max ncpus)
安装
要在您的系统上安装 gxf2bed,请按照以下步骤操作
- 获取 Rust:在 Unix 上运行
curl https://sh.rustup.rs -sSf | sh,或前往 此处 获取其他选项 - 运行
cargo install gxf2bed(确保在运行之前~/.cargo/bin已添加到您的$PATH中) - 使用
gxf2bed并提供所需的参数 - 享受吧!
构建
要从此仓库构建 gxf2bed,请执行以下操作
- 获取 Rust(如上所述)
- 运行
git clone https://github.com/alejandrogzi/gxf2bed.git && cd gxf2bed - 运行
cargo run --release -- -i <GTF/GFF> -o <BED>
容器镜像
构建开发容器镜像
- 运行
git clone https://github.com/alejandrogzi/gxf2bed.git && cd gxf2bed - 使用
start docker或systemctl start docker初始化docker - 构建镜像
docker image build --tag gxf2bed . - 运行
docker run --rm -v "[dir_where_your_gtf_is]:/dir" gxf2bed -i /dir/<GTF/GFF> -o /dir/<BED>
Conda
通过Conda使用gxf2bed,只需
conda install gxf2bed -c bioconda或conda create -n gxf2bed -c bioconda gxf2bed
基准测试 + 常见问题解答
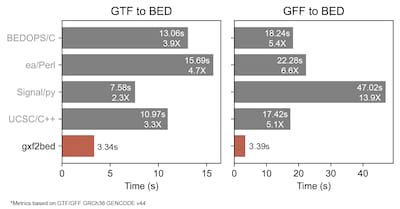
如果您在谷歌上搜索“gtf to bed”或“gff to bed”,您会发现一些关于推荐工具的帖子。其中一些工具已经过时或运行困难(甚至在Linux中),这是由于维护不当。本项目旨在提供一种简单的方法将GTF/GFF文件转换为BED结构,并完成Rust(bed2gtf、bed2gff和现在的gxf2bed)之间基因模型格式的高性能转换器。
为了测试gxf2bed的效率,我选择了4个GTF/GFF-to-BED转换器,并使用当前GRCh38 GENCODE注释(v.44),该注释有约250,000个转录本[这里显示的值是5次连续运行的平均值]。简而言之,竞争者包括
- UCSC的工具:
gtfToGenePred | genePredToBed和gffToGenePred | genePredToBed(从这里获取) - Signal & Brown:
gtf2bed.py和gff32gtf.py && gtf2bed.py(从这里获取) - ea-utils:
gtf2bed.pl和gff2gtf.pl | gtf2bed.pl(从这里获取) - BEDOPS:
gtf2bed和gtf2bed(从这里获取)
除了提供简单运行gxf2bed的方式和允许在单个转换器中集中GFF-GTF通道之外,该工具在与每种格式中的其他工具相比在计算时间上显示了显著差异。即使使用不同数量的线程组合,时间实际上也大致相同(3.5s +/- 0.4s),差异保持不变。重要的是要注意,GFF3文件所需的时间比其GTF对应物要多得多。这可能是由于需要连接两个不同的程序1)GFF-to-GTF和2)GTF-to-BED。
总的来说,gxf2bed提供了一个更简单、更快的将GTF/GFF文件转换为BED文件的方法。该工具可以为您节省至少2-3倍的时间,您以前用其他工具转换GTF文件所花费的时间,如果您想要转换GFF3文件,则可以节省5倍的时间!
依赖关系
~4–15MB
~140K SLoC