2 个不稳定版本
| 0.2.1 | 2024年1月2日 |
|---|---|
| 0.1.0 | 2023年11月2日 |
#321 在 科学
750KB
338 行
noel
使用Rust编写的超快GTF/GFF基于基因的非重叠外显子长度计算器(noel)。
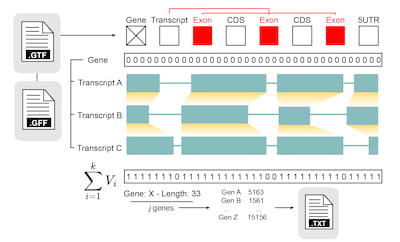
接收GTF/GFF文件,输出包含非重叠外显子长度的.txt文件。
使用方法
Usage: noel --i <GTF/GFF> --o <OUTPUT>
Arguments:
--i <GTF/GFF>: GTF/GFF file
--o <OUTPUT>: .txt file
Options:
--help: print help
--version: print version
crate: https://crates.io/crates/noel
安装
在您的系统上安装noel,请按照以下步骤操作
- 下载rust:在Unix上使用
curl https://sh.rustup.rs -sSf | sh,或访问此处获取其他选项 - 运行
cargo install noel(确保在运行之前将~/.cargo/bin添加到您的$PATH中) - 使用
noel和所需的参数
构建
要从此仓库构建noel,请执行以下操作
- 获取rust(如上所述)
- 运行
git clone https://github.com/alejandrogzi/noel.git && cd noel - 运行
cargo run --release <GTF/GFF> <OUTPUT>(参数是位置参数,因此您无需指定 --i/--o)
库
要将noel作为库包含到您的项目中,请按照以下步骤操作
-
在您的
Cargo.toml文件中包含以下代码或在命令行中运行以下命令:cargo add noel:include = 0.2.0或noel = "*"在[dependencies]下 -
库的名称是
noel,要使用它只需编写use noel::{noel, noel_reader};或者
use noel::*; -
调用
let exons: HashMap<String, Vec<(u32, u32)>> = noel_reader(input: &PathBuf)? let lengths: Vec<(String, u32)> = noel(exons) -
您将获得一个 HashMap,其中每个基因名称(gene_id)是它的长度的键
[("ENSG00000261469": 533), ("ENSG00000150990": 6908), ("ENSG00000136490": 4751), ("ENSG00000290760": 801)]
基准测试
有许多开源的工具/软件/脚本可以计算非重叠外显子长度,例如:Kooi [1]、Sun [2] 和 Slowikowski [3, 4] 脚本,以及 gtftools (-l 标志) [5]。非重叠外显子长度计算器(NOEL;简称 "noel"),作为一种新型工具,由于它的卓越性能而优于上述软件。
为了评估 noel 的效率和测试其他可用脚本/工具的功能,我使用了基于 5 次连续运行的运行时间和内存使用估计。这次评估集中在两种主要的基因注释格式上:GTF 和 GFF。然而,值得注意的是,只有三个工具能够处理 GFF 文件:Slowikowski、Sun*(以下描述)和 noel。在每次批量运行之前,我首先修改了每个脚本以使其对 CLI 响应。此外,我还编辑了 Sun 的脚本,通过更改正则表达式模式来使其能够处理 GFF 输入。没有应用与性能提升相关的更改或破坏性结构修改。
最后,为了评估排名靠前的工具(Sun、gtftools 和 noel)的输出一致性,使用了三个物种:Homo sapiens(GRCh38,GENCODE 44)、Canis lupus familiaris(ROS_Cfam_1.0,Ensembl 110)和 Mus musculus(GRCm39,GENCODE M33)。
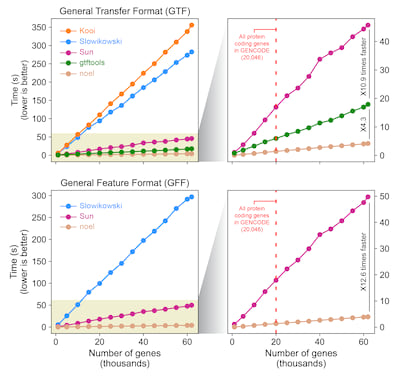
计算非重叠外显子长度的不同方法导致了运行时间的明显差异。当使用 GTF 文件时,Kooi 和 Slowikowski 脚本是最后排名的(GENCODE 44 大于 250 秒)和 Slowikowski 仅适用于 GFF 文件(GENCODE 44 大约 300 秒);Sun、gtftools 和 noel 是最有效的方法(GENCODE 44 小于 50 秒)。在分析这些排名靠前的工具时,可以迅速感知到 noel 在其竞争对手中的优势。与 gtftools(4.2 秒 vs 17.9 秒,快 4.3 倍)和 Sun 的脚本(4.2 秒 vs 45.7 秒,快 10.9 倍)相比,noel 在 GTF 文件上实现了明显更快的计算时间。另一方面,noel 的计算速度比 Sun 的脚本快 12.6 倍(3.9 秒 vs 49.7 秒)。
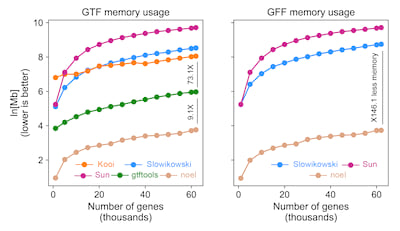
当基于 GTF 文件检查内存使用估计时,可以看到类似的模式。可以识别出三种不同的工具组:高内存消耗工具(Sun、Slowikowski 和 Kooi)、中等内存使用工具(gtftools)和最内存高效的选项(noel)。在这里,noel 与 gtftools(42.9 Mb vs 391.8 Mb,少 9.1 倍)和 Kooi(42.9 Mb vs 3.1 Gb,少 73.1 倍)相比,内存使用量明显较低。另一方面,noel 与 Slowikowski(62,700 个基因)相比,内存使用量降低了惊人的 146.1 倍。
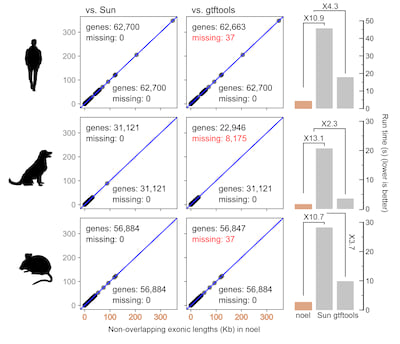
包括 Sun、gtftools 和 noel 在内的排名靠前的工具的输出比较,为每个物种提供了稳定的配对估计,结果相关性很高(R = 0.99)。值得注意的是,noel 和 Sun 的脚本在所有测试的注释模型中对每个基因都表现出一对一的对应关系。相比之下,gtftools 在处理基因时表现出局限性,在人类和鼠标模型中略有不足(分别为 0.05% 和 0.06%),在狗模型中则有更显著的不足(26%)。此外,noel 在鼠标和狗模型中优于其他工具,显著提高了运行效率,速度至少提高了 2.3 倍。
基于对现有脚本/软件计算非重叠外显子长度的比较分析以及对noel的分析,可以看出这个工具代表了一个显著的改进。这些发现揭示了noel作为快速、高效自动化非重叠外显子长度计算的有价值资源的潜力。
参考文献
[1] https://www.biostars.org/p/83901/
[2] https://gist.github.com/jsun/aeca04ee2c5b5cc53ad795b660edd6c3
[3] https://gist.github.com/slowkow/8101481
[4] https://gist.github.com/slowkow/8101509#file-coding_lengths-py
[5] 李红东,林翠香,郑剑涛,GTFtools:一个用于分析基因模型各种特征的软件包,生物信息学,2022年10月15日第38卷第20期,第4806-4808页,https://doi.org/10.1093/bioinformatics/btac561
依赖关系
~2.4–3MB
~61K SLoC