23 个版本
使用旧的 Rust 2015
| 0.1.22 | 2023年2月24日 |
|---|---|
| 0.1.20 | 2023年1月29日 |
| 0.1.18 | 2022年12月27日 |
| 0.1.17 | 2022年11月23日 |
| 0.1.0 | 2020年7月13日 |
#78 in 生物学
500KB
2.5K SLoC
RNA 结构比对快速概率推理引擎
安装
此项目是用 Rust 编写的,一种系统编程语言。您需要安装 Rust 组件,即 rustc(Rust 编译器)、cargo(Rust 包管理器)和 Rust 标准库。访问 Rust 网站 了解更多关于 Rust 的信息。您可以使用以下单行命令安装 Rust 组件:
curl --proto '=https' --tlsv1.2 -sSf https://sh.rustup.rs | sh
Rustup 安装上述组件并允许轻松切换正在使用的编译器。您可以使用以下命令安装 ConsProb
# AVX, SSE, and MMX enabled for rustc
# Another example: RUSTFLAGS='--emit asm -C target-feature=+avx2 -C target-feature=+ssse3 -C target-feature=+mmx -C target-feature=+fma'
RUSTFLAGS='--emit asm -C target-feature=+avx -C target-feature=+ssse3 -C target-feature=+mmx' \
cargo install consprob
检查您是否已正确安装 ConsProb
# Its available command options will be displayed
consprob
您可以使用准备好的样本 tRNA 测试集运行 ConsProb
git clone https://github.com/heartsh/consprob \
&& cd consprob
cargo test --release
# The below command requires Gnuplot (http://www.gnuplot.info)
# Benchmark results will be found at "./target/criterion/report/index.html"
cargo bench
RNA 结构上下文轮廓的先进计算
测量每个 RNA 核苷酸的结构上下文轮廓(即每个核苷酸在每个结构上下文类型中的后验概率)对围绕功能非编码 RNA 的各种结构分析有益。例如,CapR 在 RNA 二级结构上计算 RNA 结构上下文轮廓,区分以下结构上下文类型:(1)发夹环中的非配对,(2)碱基配对,(3)突起环中的非配对,(4)内部环中的非配对,(5)多环中的非配对,以及(6)外环中的非配对
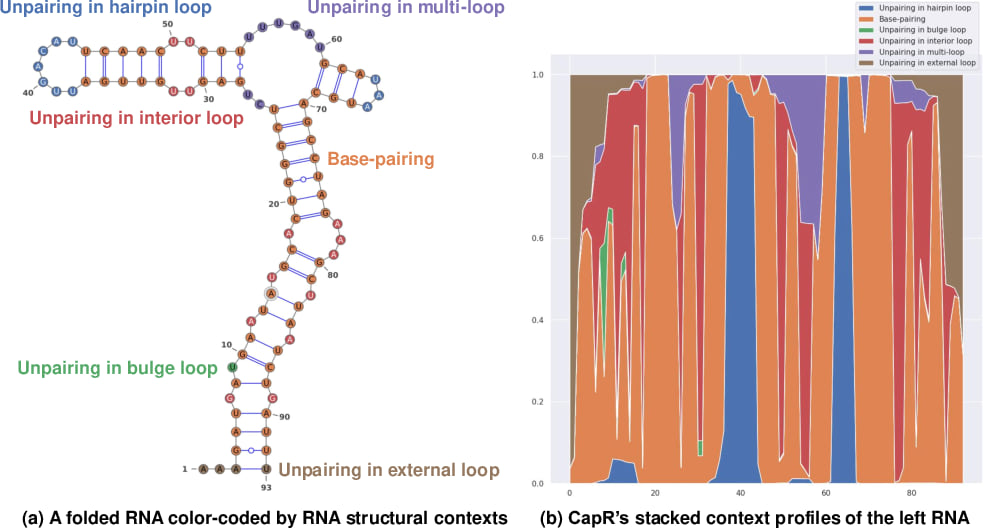 |
|---|
尊重 CapR,ConsProb 提供了 RNA 结构比对上平均结构上下文轮廓的计算,区分上述结构上下文类型。技术上,ConsProb 计算 RNA 成对结构比对上每个核苷酸对的结构上下文轮廓,并将此成对上下文轮廓平均到每个 RNA 同源物上,对可用的 RNA 同源物进行边缘化。ConsProb 的上下文轮廓计算在 ConsProb 的论文中没有描述。但是,您可以轻松地通过定制 ConsProb 的主内-外算法来计算后验核苷酸对匹配概率,因为 CapR 基于McCaskill算法。以下是 ConsProb 的平均上下文轮廓示例
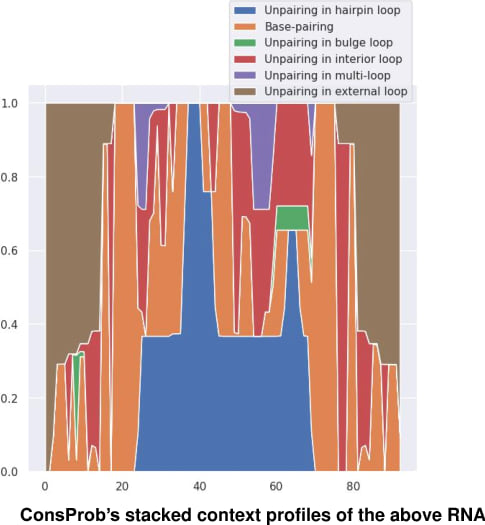 |
|---|
Docker 沙盒 
我提供基于Docker的RNA软件及其说明的实验平台我的实验平台,以便轻松回放我的计算实验。
方法摘要
LocARNA-P可以在RNA成对结构比对上计算后验核苷酸配对匹配概率。然而,LocARNA-P通过利用RNA二级结构上的后验核苷酸配对概率来简化可能的成对结构比对的评分。换句话说,LocARNA-P不使用与许多RNA折叠方法相同的评分复杂度来评分可能的成对结构比对。更具体地说,许多RNA折叠方法(如RNAfold)通过区分RNA环结构来评分可能的RNA二级结构,而许多基于结构比对的方法(如LocARNA-P)则忽略RNA环结构来评分可能的成对结构比对。作为这些基于结构比对方法的对立面,我在本存储库中实现了ConsProb。区分RNA环结构,ConsProb快速估计各种成对后验概率,包括后验核苷酸配对匹配概率。ConsProb将这些估计的成对概率总结为平均概率一致性,将多个RNA同源物归一化到每个RNA同源物。
作者
许可
版权所有(c)2018 Heartsh
许可协议:MIT许可。
依赖项
~58MB
~1.5M SLoC