1 个不稳定版本
| 0.1.4 | 2024年3月12日 |
|---|
#64 在 生物学 中
5MB
2K SLoC
SeqSizzle 是一个具有模糊匹配功能的快速查看器,允许不同适配器以不同颜色显示。
最新预构建的二进制文件
使用方法
./seqsizzle -h:
Usage: seqsizzle [OPTIONS] <FILE> [COMMAND]
Commands:
summarize Summarize the reads with patterns specified by the --patterns argument or the adapter flags. Make sure you supply the flags BEFORE the subcommand, e.g. `./SeqSizzle my.fastq -p my_patterns.csv --adapter-3p summarize`. '..' indicats unmatched regions of positive length, '-' indicates the patterns are overlapped, print the number of reads that match each pattern combination in TSV format. To be moved to the UI in the future
help Print this message or the help of the given subcommand(s)
Arguments:
<FILE> The FASTQ file to view
Options:
--adapter-3p
Start with 10x 3' kit adaptors:
- Patrial Read1: CTACACGACGCTCTTCCGATCT (and reverse complement)
- Partial TSO: AGATCGGAAGAGCGTCGTGTAG (and reverse complement)
- Poly(>10)A/T
--adapter-5p
Start with 10x 5' kit adaptors
- Patrial Read1: CTACACGACGCTCTTCCGATCT (and reverse complement)
- TSO: TTTCTTATATGGG (and reverse complement)
- Patrial Read2: AGATCGGAAGAGCACACGTCTGAA (and reverse complement)
- Poly(>10)A/T
-p, --patterns <PATTERNS_PATH>
Start with patterns from a CSV file
Must have the following header:
pattern,color,editdistance,comment
-s, --save-patterns <SAVE_PATTERNS_PATH>
Save the search panel to a CSV file before quitting. To be removed in the future since you can now hit Ctrl-S in the search panel to save the patterns
-h, --help
Print help
-V, --version
Print version
导航
查看模式
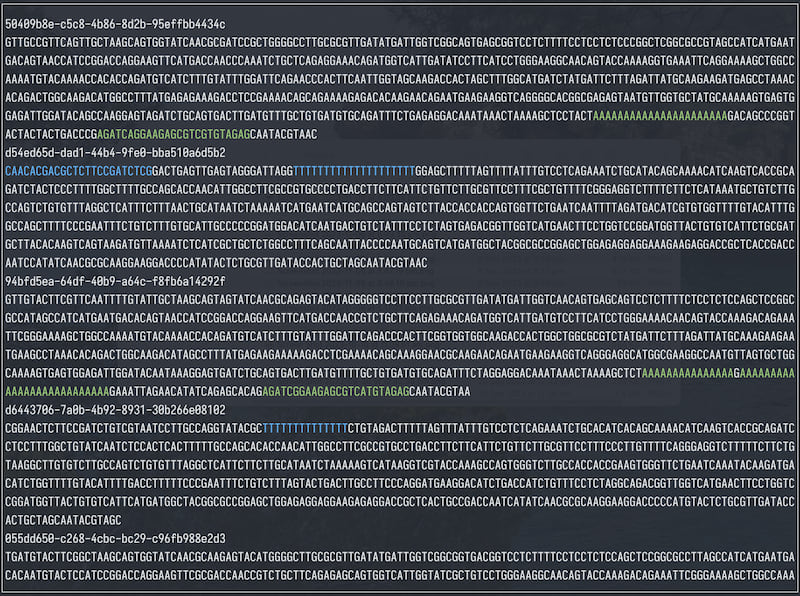 向上/向下箭头(或
向上/向下箭头(或 j / k)滚动一行,Ctrl+U / Ctrl+D 滚动半屏。
/(或 Ctrl+F)切换搜索面板,q 退出
搜索面板模式
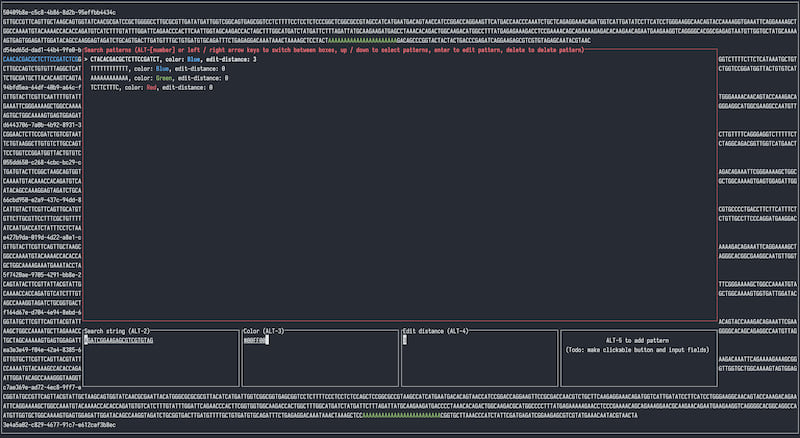 左/右箭头(或 Tab / Shift-Tab)在不同的输入字段和模式列表之间循环。
左/右箭头(或 Tab / Shift-Tab)在不同的输入字段和模式列表之间循环。
当在模式列表字段中时,上/下箭头循环通过模式,Backspace(或 Delete,d)删除所选模式,Return 将模式弹出至输入字段进行编辑。
Return 将当前输入添加到搜索模式列表(当聚焦于任何输入框时,而不是模式列表)。
使用 Shift + 箭头键在输入字段内移动光标(因为单独的箭头键绑定到循环输入字段)。
/ 或 Esc 关闭搜索面板。
路线图
功能
- Gzip(
fastq.gz)支持 - 通过匹配过滤读取
- 匹配读取计数
用户界面
- 使搜索面板中的元素可点击,尝试在 ratatui 代码库 中讨论的实现
杂项
- 单元测试
依赖
~35–47MB
~831K SLoC