11个版本
| 0.5.1 | 2024年6月23日 |
|---|---|
| 0.5.0 | 2023年8月5日 |
| 0.4.0 | 2023年5月4日 |
| 0.3.0 | 2022年11月16日 |
| 0.1.4 | 2021年7月7日 |
#13 in 生物学
445KB
6.5K SLoC
block aligner
使用自适应块算法计算全局和X-drop斜率缺口惩罚序列到序列或序列到轮廓对齐的SIMD加速库。
有关算法的更多信息及其与其他算法的比较,请参阅生物信息学论文此处。
如果您发现Block Aligner很有用,请引用
@article{liu2023block,
title={{Block Aligner}: an adaptive {SIMD}-accelerated aligner for sequences and position-specific scoring matrices},
author={Liu, Daniel and Steinegger, Martin},
journal={Bioinformatics},
volume={39},
number={8},
pages={btad487},
year={2023},
publisher={Oxford University Press}
}
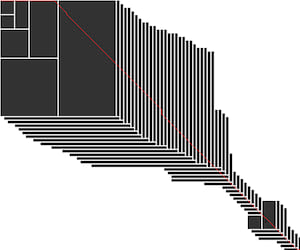
示例
use block_aligner::{cigar::*, scan_block::*, scores::*};
let min_block_size = 32;
let max_block_size = 256;
// A gap of length n will cost: open + extend * (n - 1)
let gaps = Gaps { open: -2, extend: -1 };
// Note that PaddedBytes, Block, and Cigar can be initialized with sequence length
// and block size upper bounds and be reused later for shorter sequences, to avoid
// repeated allocations.
let r = PaddedBytes::from_bytes::<NucMatrix>(b"TTAAAAAAATTTTTTTTTTTT", max_block_size);
let q = PaddedBytes::from_bytes::<NucMatrix>(b"TTTTTTTTAAAAAAATTTTTTTTT", max_block_size);
// Align with traceback, but no X-drop threshold (global alignment).
let mut a = Block::<true, false>::new(q.len(), r.len(), max_block_size);
a.align(&q, &r, &NW1, gaps, min_block_size..=max_block_size, 0);
let res = a.res();
assert_eq!(res, AlignResult { score: 7, query_idx: 24, reference_idx: 21 });
let mut cigar = Cigar::new(res.query_idx, res.reference_idx);
// Compute traceback and resolve =/X (matches/mismatches).
a.trace().cigar_eq(&q, &r, res.query_idx, res.reference_idx, &mut cigar);
assert_eq!(cigar.to_string(), "2=6I16=3D");
有关详细的API信息,请参阅文档。
以下是一个显示Block Aligner支持的对齐类型的图解
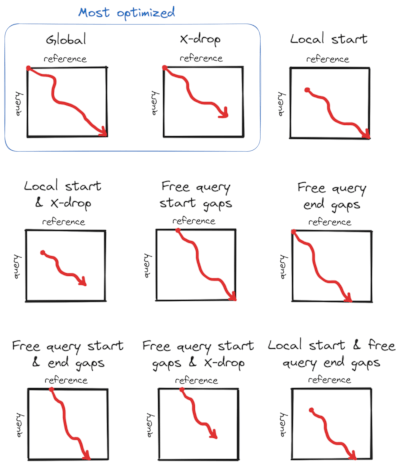
算法
Block aligner通过动态规划提供了一种新的高效方法来计算蛋白质、DNA序列和字节字符串的成对对齐。Block aligner还支持将序列与轮廓对齐,轮廓是位置特定的得分矩阵和位置特定的缺口开放成本。
它通过计算一个小正方形块中的得分,并根据块边缘的得分以贪婪的方式向下或向右移动来实现。这种动态方法与以前的方法相比,计算出的块区域要小得多,尽管牺牲了一些精度。块还可以回到先前的最佳检查点并变大,以处理具有大缺口的困难区域。当检测到不需要大块时,块大小还可以动态缩小。块增长和缩小都基于启发式方法。
通过以速度为代价牺牲一些精度,block aligner能够高效地处理各种得分矩阵并适应具有不同序列同源性的序列。实际上,它在各种蛋白质和核酸序列上仍然非常准确。
块对齐器旨在利用现代CPU上的SIMD并行性。目前支持SSE2(128位向量)、AVX2(256位向量)、Neon(128位向量)和WASM SIMD(128位向量)。对于分数计算,使用16位分数值(通道)和每个块32位偏移量。
当设置最小和最大块大小为相同值时,块对齐器的行为类似于(自适应)带状对齐器。
调整块大小
对于长、嘈杂的Nanopore读取,最小块大小为序列长度的约1%,最大块大小为序列长度的约10%表现良好(用高达约50kbps的读取进行测试)。对于蛋白质,最小块大小为32,最大块大小为256表现良好。建议使用至少32的最小块大小。不建议使用大于2^14 = 16384的最大块大小。库中包含一个percent_len函数,可以计算基于这些推荐的序列长度的百分比。如果对齐分数饱和(分数太大),则使用更小的块大小。请告诉我块对齐器在您的数据上的表现如何!
安装
此库可以在稳定和夜间Rust通道上使用。夜间通道需要运行测试和基准测试。此外,测试和基准测试需要在Linux或MacOS上运行。
要将此用作Rust项目中的crate,请将以下内容添加到您的Cargo.toml
[dependencies]
block-aligner = { version = "0.5", features = ["simd_avx2"] }
使用simd_sse2、simd_neon或simd_wasm功能标志分别启用x86 SSE2、ARM Neon或WASM SIMD支持。确保启用的功能和平台支持正确,因为此库不会自动检测支持的SIMD指令集。有关如何为具有相同依赖项的不同平台指定不同功能的更多信息,请参阅这里。以下是一个简单示例
[target.'cfg(target_arch = "x86_64")'.dependencies]
block-aligner = { version = "0.5", features = ["simd_avx2"] }
[target.'cfg(target_arch = "aarch64")'.dependencies]
block-aligner = { version = "0.5", features = ["simd_neon"] }
对于开发、测试或使用C API,您应该克隆此存储库并使用Rust夜间版本。通常,在构建时,您需要通过命令行指定正确的功能标志。
对于x86 AVX2
cargo build --features simd_avx2 --release
对于x86 SSE2
cargo build --features simd_sse2 --release
对于ARM Neon
cargo build --target=aarch64-unknown-linux-gnu --features simd_neon --release
对于WASM SIMD
cargo build --target=wasm32-wasi --features simd_wasm --release
要运行WASM程序,您需要安装wasmtime并在您的$PATH上。
C API
块对齐器有C绑定。有关如何使用它们的更多信息,请参阅C README。查看3di分支以获取使用块对齐器在C中进行局部对齐的示例,以及块对齐器修改以支持使用氨基酸3D相互作用(3Di)信息对齐。
改进块对齐器
在对齐过程中,每个步骤需要做出三个决定(使用启发式方法)
- 是否增长块大小
- 是否缩小块大小
- 是否向右或向下移动
块对齐器使用简单的贪婪启发式方法来做出这些决定,这些方法容易评估。这里可能有很大的改进空间!也许种子?神经网络模型?
为了尝试您的想法,请查看代码注释之后的代码 // TODO: 更好的启发式方法?,位于 src/scan_block.rs 中(根据您的更改,您可能还需要修改代码的其他部分)。如果您正在尝试新想法,请告诉我!
以下大多数说明用于基准测试和测试块对齐器。
数据
一些测试和基准测试使用了Illumina/Nanopore(DNA)、Uniclust30(蛋白质)和SCOP(蛋白质轮廓)数据。您需要按照 数据README 中的说明下载它们。
测试
运行 scripts/test_avx2.sh 或 scripts/test_wasm.sh 来运行测试。当提交推送到此仓库时,CI将运行这些测试。更多测试和评估脚本可在 scripts 目录中找到。
对于调试,存在一个 debug 功能标志,在运行时会打印出大量有关对齐器内部状态的有用信息。还有一个名为 debug_size 的功能标志,会在块增长后打印其大小。要手动检查对齐,请用两个序列作为参数运行 scripts/debug_avx2.sh。
文档
运行 scripts/doc_avx2.sh 或 scripts/doc_wasm.sh 以在本地构建文档。
基准测试
运行 scripts/bench_avx2.sh 或 scripts/bench_wasm.sh 以进行基本基准测试。在 scripts 目录中可以找到在真实数据上可运行的基准测试脚本。基准测试的实际实现大多位于 examples 目录中。
数据分析与可视化
使用 vis/ 目录中的Jupyter笔记本收集数据和绘图。运行整个笔记本的更简单方法是运行 vis/run_vis.sh 脚本。这重复了手册中的实验。
使用MacOS Instruments进行性能分析
使用
brew install cargo-instruments
RUSTFLAGS="-g" cargo instruments --example profile --release --features simd_avx2 --open
使用LLVM-MCA分析性能
使用
scripts/build_ir_asm.sh
生成汇编输出并运行LLVM-MCA。
查看汇编
使用 scripts/build_ir_asm.sh、在二进制文件上使用 objdump -d(在某些情况下可以避免重新编译代码),或使用更高级的工具(如Ghidra,它也有反编译器)。
比较(相对不常用)
对 Hajime Suzuki 的自适应带状基准代码和差异递归基准代码进行了编辑。这些编辑分别可在 此处 和 此处 找到。转到那些仓库,然后按照安装和运行代码的说明进行操作。
如果您运行这些仓库中的脚本以比较不同算法产生的分数,您应该会生成 .tsv 文件。然后,在此仓库目录中运行
scripts/compare_avx2.sh /path/to/file.tsv 50
以获取比较。X-drop阈值位于路径之后。
旧想法和历史
请参阅 想法 文件。