11 个不稳定版本 (5 个重大变更)
| 0.8.3 | 2024 年 7 月 18 日 |
|---|---|
| 0.7.0 | 2024 年 6 月 10 日 |
| 0.6.0 | 2023 年 10 月 5 日 |
| 0.5.1 | 2023 年 3 月 13 日 |
| 0.3.0 | 2021 年 7 月 31 日 |
在 模拟 中排名 3
每月下载量 120
115KB
688 行
rebop
rebop 是一个用于良好混合的化学反应网络的快速随机模拟器。
性能和用户体验被非常重视。因此,提供了两个独立的 API 来描述和模拟反应网络
- 一个由宏实现的 DSL,由
define_system实现,通常效率最高,但需要编译 Rust 程序; - 一个由模块
gillespie实现的基于函数的 API,也可以通过 Python 绑定访问。这个 API 不需要 Rust 编译,允许在运行时定义系统。它的速度通常比宏 DSL 慢 2 到 3 倍,但仍然比其他所有尝试过的软件快。
宏 DSL
它目前仅支持由质量作用定律定义的反应速率。以下宏定义了一个二聚化反应网络
use rebop::define_system;
define_system! {
r_tx r_tl r_dim r_decay_mRNA r_decay_prot;
Dimers { gene, mRNA, protein, dimer }
transcription : gene => gene + mRNA @ r_tx
translation : mRNA => mRNA + protein @ r_tl
dimerization : 2 protein => dimer @ r_dim
decay_mRNA : mRNA => @ r_decay_mRNA
decay_protein : protein => @ r_decay_prot
}
要模拟系统,请将此定义放入 Rust 代码文件中,实例化问题,设置参数、初始值,并启动模拟
let mut problem = Dimers::new();
problem.r_tx = 25.0;
problem.r_tl = 1000.0;
problem.r_dim = 0.001;
problem.r_decay_mRNA = 0.1;
problem.r_decay_prot = 1.0;
problem.gene = 1;
problem.advance_until(1.0);
println!("t = {}: dimer = {}", problem.t, problem.dimer);
或者对于经典的 SIR 例子
use rebop::define_system;
define_system! {
r_inf r_heal;
SIR { S, I, R }
infection : S + I => 2 I @ r_inf
healing : I => R @ r_heal
}
fn main() {
let mut problem = SIR::new();
problem.r_inf = 1e-4;
problem.r_heal = 0.01;
problem.S = 999;
problem.I = 1;
println!("time,S,I,R");
for t in 0..250 {
problem.advance_until(t as f64);
println!("{},{},{},{}", problem.t, problem.S, problem.I, problem.R);
}
}
可以产生类似这样的输出
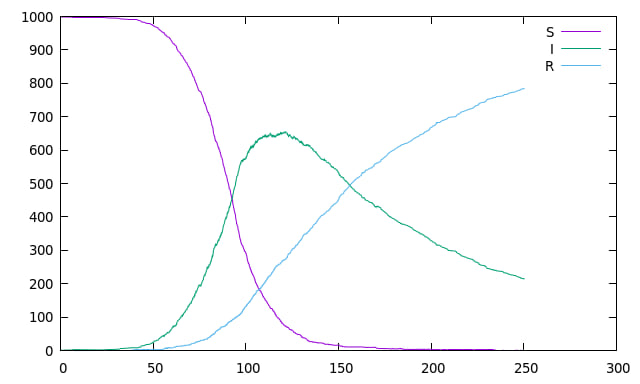
Python 绑定
此 API 通过 Python 绑定而更加出色,允许用户轻松定义模型
import rebop
sir = rebop.Gillespie()
sir.add_reaction(1e-4, ['S', 'I'], ['I', 'I'])
sir.add_reaction(0.01, ['I'], ['R'])
print(sir)
ds = sir.run({'S': 999, 'I': 1}, tmax=250, nb_steps=250)
您可以通过从 PyPI 安装 rebop 来测试此代码,使用 pip install rebop。要从源码构建 Python 绑定,最简单的方法是克隆此 Git 仓库并使用 maturin develop。
传统 API
Python 包底层的基于函数的 API 也可以从 Rust 访问,如果您想在运行时定义模型(而不是使用上述宏 DSL 在编译时定义)。SIR 模型定义为
use rebop::gillespie::{Gillespie, Rate};
let mut sir = Gillespie::new([999, 1, 0]);
// [ S, I, R]
// S + I => 2 I with rate 1e-4
sir.add_reaction(Rate::lma(1e-4, [1, 1, 0]), [-1, 1, 0]);
// I => R with rate 0.01
sir.add_reaction(Rate::lma(0.01, [0, 1, 0]), [0, -1, 1]);
println!("time,S,I,R");
for t in 0..250 {
sir.advance_until(t as f64);
println!("{},{},{},{}", sir.get_time(), sir.get_species(0), sir.get_species(1), sir.get_species(2));
}
性能
性能被非常重视,因此 rebop 在性能上优于我们尝试过的每个其他软件包和编程语言。
免责声明:该软件目前包含的功能远超rebop(例如空间模型、自定义反应速率等)。其中一些功能可能需要它们在速度上做出妥协。此外,尽管我们尽量保持比较公平,但有些返回的数据过多或过少,或者将它们写入磁盘。我们试图接近所有这些程序的基本线如下: 模型刚刚修改,我们想模拟它N次,并打印等间距的测量点。这意味着,如果适用,我们始终包括初始化或(重新)编译时间。我们认为,这是研究模型的科研人员最典型的使用场景。这种方法可以记录初始化时间(y截距)和每次模拟的模拟时间(斜率)。
在许多玩具示例上进行的小型基准测试被追踪以指导开发。为了与其他软件比较性能,我们使用了一个低中尺寸的真实世界模型(9种物种和16个反应):Vilar振荡器(《遗传振荡器的噪声抗性机制》,Vilar等人,PNAS 2002)。在这里,我们从这个模型t=0模拟到t=200,以时间单位的时间间隔报告状态。
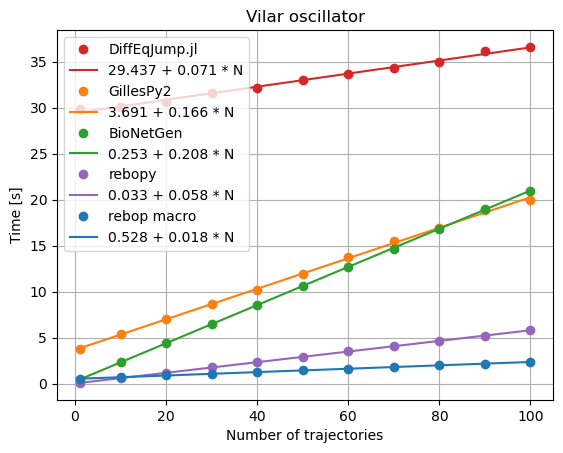
我们可以看到,rebop的宏DSL在每次模拟时间和包含编译时间方面都是最快的。其次是rebop的传统API,通过Python绑定方便地调用。
即将到来的功能
- 隔室体积
- 任意反应速率
- 其他SSA算法
- tau跳跃
- 自适应tau跳跃
- 混合模型(连续和离散)
- SBML
- 命令行界面
- 参数估计
- 局部敏感性分析
- 并行化
可能不会到来的功能
- 事件
- 空间(反应-扩散系统)
- 规则建模
基准测试想法
- DSMTS
- 纯解耦指数
- 环
- 切换开关
- LacZ,LacY/LacZ(来自STOCKS)
- Lotka Volterra,Michaelis-Menten,网络(来自StochSim)
- G蛋白(来自SimBiology)
- Brusselator / Oregonator(来自Cellware)
- GAL,repressilator(来自Dizzy)
类似的软件
维护
- GillesPy2
- STEPS
- SimBiology
- Copasi
- BioNetGen
- VCell
- Smoldyn
- KaSim
- StochPy
- BioSimulator.jl
- DiffEqJump.jl
- Gillespie.jl
- GillespieSSA2
- Cayenne
看起来未维护
许可:MIT
依赖项
~3.5MB
~68K SLoC