13个版本
| 0.10.0 | 2023年3月11日 |
|---|---|
| 0.9.0 | 2022年5月9日 |
| 0.8.6 | 2022年2月27日 |
| 0.8.2 | 2021年10月5日 |
| 0.1.1 | 2020年3月12日 |
203 在 科学
42 每月下载量
94KB
1.5K SLoC
nanoq 
纳米孔读取的超快速质量控制和总结报告
概述
v0.10.0
目的
Nanoq实现了对高通量纳米孔读取的超快速读取过滤和总结报告。
引用
如果您在研究中使用nanoq,我们将非常感谢您的引用。有关如何在使用nanoq用于工业应用时回馈社区的某些建议,请参阅此处 🙏
Steinig和Coin (2022)。Nanoq:纳米孔读取的超快速质量控制。开源软件杂志,7(69),2991,https://doi.org/10.21105/joss.02991
性能
Nanoq对于小型数据集的摘要统计(例如,Zymo - 100,000个读取)与seqtk-fqchk一样快,并在大型数据集(例如,Zymo - 3.5百万读取,1.3x - 1.5x)上略快。在fast模式下(没有质量分数),对于摘要统计和其他常用的读取摘要或过滤方法(高达297x-442x),nanoq比rust-bio-tools和seqkit stats快2-3倍。内存消耗一致,并且往往低于其他工具(~5-10x)。
测试
Nanoq附带高测试覆盖率,让您放心。
cargo test
安装
Cargo
cargo install nanoq
Conda
显式版本(由于某些原因默认为旧版本)
conda install -c conda-forge -c bioconda nanoq=0.10.0
二进制文件
最新版本附带了Linux和MacOS的预编译二进制文件。
VERSION=0.10.0
RELEASE=nanoq-${VERSION}-x86_64-unknown-linux-musl.tar.gz
wget https://github.com/esteinig/nanoq/releases/download/${VERSION}/${RELEASE}
tar xf nanoq-${VERSION}-x86_64-unknown-linux-musl.tar.gz
nanoq-${VERSION}-x86_64-unknown-linux-musl/nanoq -h
用法
Nanoq 接受格式为 fast{a,q}.{gz,bz2,xz} 的文件(-i)或流(stdin),并将读取结果输出到文件(-o)或流(stdout)。
nanoq -i test.fq.gz -o reads.fq
cat test.fq.gz | nanoq > reads.fq
读取过滤器
读取可以通过最小读取长度(-l)、最大读取长度(-m)、最小平均读取质量(-q)或最大平均读取质量(-w)进行过滤。
nanoq -i test.fq -l 1000 -m 10000 -q 10 -w 15 > reads.fq
读取修剪
可以从读取的起始(-S)或结束(-E)处修剪固定数量的碱基。
nanoq -i test.fq -S 100 -E 100 > reads.fq
读取报告
当使用统计标志(-s,报告到 stdout,不向 stdout 输出读取)或指定报告文件(-r)时,将生成读取摘要。
nanoq -i test.fq -s
nanoq -i test.fq -r report.txt > reads.fq
有关报告类型和配置,请参阅输出部分。
快速模式
⚠️ 使用快速模式
-f时,不计算读取质量得分(质量字段输出:NaN)
读取质量可以排除在过滤器和统计之外,以加快读取迭代(-f)。
nanoq -i test.fq.gz -f -s
压缩
输出压缩由文件扩展名推断(gz、bz2、lzma)。
nanoq -i test.fq -o reads.fq.gz
可以使用 -O 和 -c 手动指定输出压缩。
nanoq -i test.fq -O g -c 9 -o reads.fq.gz
在线运行
Nanoq 可用于检查正在进行的测序运行和条形码样本。
find /data/nanopore/run -name "*.fastq" -print0 | xargs -0 cat | nanoq -s
for i in {01..12}; do
find /data/nanopore/run -name barcode${i}.fastq -print0 | xargs -0 cat | nanoq -s
done
参数
nanoq 0.10.0
Filters and summary reports for nanopore reads
USAGE:
nanoq [FLAGS] [OPTIONS]
FLAGS:
-f, --fast Ignore quality values if present
-h, --help Prints help information
-H, --header Header for summary output
-j, --json Summary report in JSON format
-s, --stats Summary report only [stdout]
-V, --version Prints version information
-v, --verbose Verbose output statistics [multiple, up to -vvv]
OPTIONS:
-c, --compress-level <1-9> Compression level to use if compressing output [default: 6]
-i, --input <input> Fast{a,q}.{gz,xz,bz}, stdin if not present
-m, --max-len <INT> Maximum read length filter (bp) [default: 0]
-w, --max-qual <FLOAT> Maximum average read quality filter (Q) [default: 0]
-l, --min-len <INT> Minimum read length filter (bp) [default: 0]
-q, --min-qual <FLOAT> Minimum average read quality filter (Q) [default: 0]
-o, --output <output> Output filepath, stdout if not present
-O, --output-type <u|b|g|l> u: uncompressed; b: Bzip2; g: Gzip; l: Lzma
-r, --report <FILE> Summary read statistics report output file
-t, --top <INT> Number of top reads in verbose summary [default: 5]
-L, --read-lengths <FILE> Output read lengths of surviving reads to file
-Q, --read-qualities <FILE> Output read qualities of surviving reads to file
-S, --trim-start <INT> Trim bases from the start of each read [default: 0]
-E, --trim-end <INT> Trim bases from the end of each read [default: 0]
输出
读取长度和质量
可以输出存活读取的长度(--read-lengths/-L)和质量(--read-qualities/-Q)的文件。
nanoq -i test.fq -Q rq.txt -L rl.txt > reads.fq
摘要报告
使用 --report/-r 显式将摘要报告输出到文件。
nanoq -i test.fq -r report.txt > reads.fq
nanoq -i test.fq -r report.txt -s
使用 --stats/-s 标志时,将抑制读取输出,并将摘要定向到 stdout。
nanoq -i test.fq -s > report.txt
默认情况下,报告格式是最小的。
100000 400398234 5154 44888 5 4003 3256 8.90 9.49
- 读取数量
- 碱基对数量
- N50 读取长度
- 最长读取
- 最短读取
- 平均读取长度
- 中值读取长度
- 平均读取质量
- 中值读取质量
可以使用 -H 标志添加可机器读取的标题。
nanoq -i test.fq -s -H
使用多个 - 标志(最多 -vvv)可以获得类似 NanoStat 的扩展摘要,包括顶级(-)读取长度和质量
-- 详细读取摘要(如下面的顶部块)-- 与-类似,带有读取长度和/或质量阈值-- 与-类似,带有顶级排序的读取长度和/或质量
nanoq -i test.fq -f -s -t 5 -vvv
Nanoq Read Summary
====================
Number of reads: 100000
Number of bases: 400398234
N50 read length: 5154
Longest read: 44888
Shortest read: 5
Mean read length: 4003
Median read length: 3256
Mean read quality: NaN
Median read quality: NaN
Read length thresholds (bp)
> 200 99104 99.1%
> 500 96406 96.4%
> 1000 90837 90.8%
> 2000 73579 73.6%
> 5000 25515 25.5%
> 10000 4987 05.0%
> 30000 47 00.0%
> 50000 0 00.0%
> 100000 0 00.0%
> 1000000 0 00.0%
Top ranking read lengths (bp)
1. 44888
2. 40044
3. 37441
4. 36543
5. 35630
使用 --json/-j 标志,可以将格式化的 JSON 扩展输出(相当于 -vvv)输出到 --report(-r)或 stdout(-s)。
nanoq -i test.fq --json -f -r report.json > reads.fq
nanoq -i test.fq --json -f -s > report.json
{
"reads": 100000,
"bases": 400398234,
"n50": 5154,
"longest": 44888,
"shortest": 5,
"mean_length": 4003,
"median_length": 3256,
"mean_quality": null,
"median_quality": null,
"length_thresholds": {
"200": 99104,
"500": 96406,
"1000": 90837,
"2000": 73579,
"5000": 25515,
"10000": 4987,
"30000": 47,
"50000": 0,
"100000": 0,
"1000000": 0
},
"quality_thresholds": {
"5": 0,
"7": 0,
"10": 0,
"12": 0,
"15": 0,
"20": 0,
"25": 0,
"30": 0
},
"top_lengths": [
44888, 40044, 37441, 36543, 35630
],
"top_qualities": []
}
请注意,在这个示例中未计算读取质量;因此,质量阈值全部为零。
基准测试
基准测试评估了基本读取长度过滤和偶数 Zymo 模拟社区(GridION)的摘要统计信息,并与 rust-bio-tools、seqtk fqchk、seqkit stats、NanoFilt、NanoStat 和 Filtlong 进行比较。使用 /usr/bin/time -f "%e %M" 测量完成时间和最大内存消耗,速度提升是相对于集合中最慢的命令。我们注意到,rust-bio-tools 和 seqkit stats 的摘要统计信息不计算读取质量分数,因此与 nanoq-fast 相当。
任务
stats:基本读取集摘要filter:最小读取长度过滤器(输出到/dev/null)
工具
rust-bio-tools0.28.0nanostat1.5.0nanofilt2.8.0filtlong0.2.1seqtk1.3-r126seqkit2.0.0nanoq0.8.2
用于 stats 任务的命令
nanostat(fq + fq.gz)-->NanoStat --fastq test.fq --threads 1rust-bio(fq)-->rbt sequence-stats --fastq < test.fqrust-bio(fq.gz)-->zcat test.fq.gz | rbt sequence-stats --fastqseqtk-fqchk(fq + fq.gz)-->seqtk fqchkseqkit stats(fq + fq.gz)-->seqkit stats -j1nanoq(fq + fq.gz)-->nanoq --input test.fq --statsnanoq-fast(fq + fq.gz)-->nanoq --input test.fq --stats --fast
用于 filter 任务的命令
filtlong(fq + fq.gz)-->filtlong --min_length 5000 test.fq > /dev/nullnanofilt(fq) -->NanoFilt --fastq test.fq --length 5000 > /dev/nullnanofilt(fq.gz) -->gunzip -c test.fq.gz | NanoFilt --length 5000 > /dev/nullnanoq(fq + fq.gz) -->nanoq --input test.fq --min-len 5000 > /dev/nullnanoq-fast(fq + fq.gz) -->nanoq --input test.fq --min-len 5000 --fast > /dev/null
文件
zymo.fq:未压缩(100,000 条读段,约 400 Mbp)zymo.fq.gz:压缩(100,000 条读段,约 400 Mbp)zymo.full.fq:未压缩(3,491,078 条读段,约 14 Gbp)
数据准备
wget "https://nanopore.s3.climb.ac.uk/Zymo-GridION-EVEN-BB-SN.fq.gz"
zcat Zymo-GridION-EVEN-BB-SN.fq.gz > zymo.full.fq
head -400000 zymo.full.fq > zymo.fq && gzip -k zymo.fq
消耗的实际时间和最大常驻集大小
/usr/bin/time -f "%e %M"
任务和命令执行
命令在提供的 Docker 容器中运行了 10 次重复,并且在一个挂载的基准数据卷中进行。在最终的基准测试中没有考虑每个命令的额外冷启动迭代。
for i in {1..11}; do
for f in /data/*.fq; do
/usr/bin/time -f "%e %M" nanoq -f- s -i $f 2> benchmark
tail -1 benchmark >> nanoq_stat_fq
done
done
基准结果
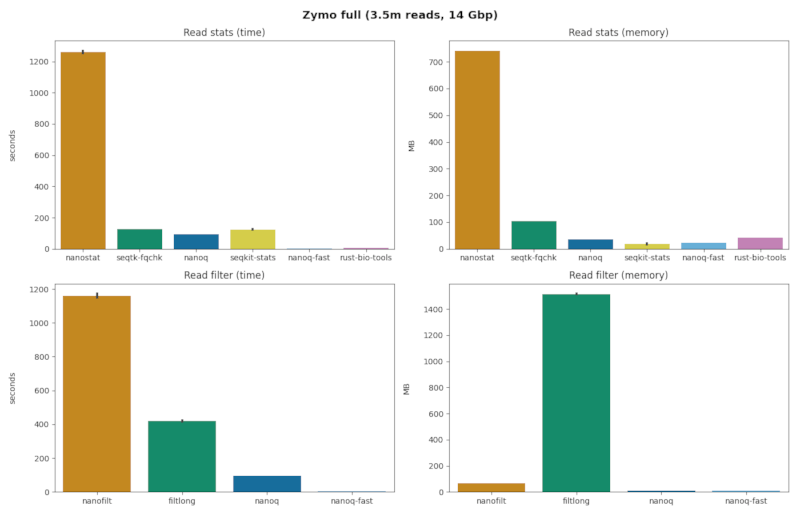
stats + zymo.full.fq
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 | 质量分数 |
|---|---|---|---|---|---|
| nanostat | 741.4 (0.09) | 1260. (13.9) | 2,770 | 01.00 x | 真 |
| seqtk-fqchk | 103.8 (0.04) | 125.9 (0.15) | 27,729 | 10.01 x | 真 |
| seqkit-stats | 18.68 (3.15) | 125.3 (0.91) | 27,861 | 10.05 x | 假 |
| nanoq | 35.83 (0.06) | 94.51 (0.43) | 36,938 | 13.34 x | 真 |
| rust-bio | 43.20 (0.08) | 06.54 (0.05) | 533,803 | 192.7 x | 假 |
| nanoq-fast | 22.18 (0.07) | 02.85 (0.02) | 1,224,939 | 442.1 x | 假 |
filter + zymo.full.fq
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 |
|---|---|---|---|---|
| nanofilt | 67.47 (0.13) | 1160. (20.2) | 3,009 | 01.00 x |
| filtlong | 1516. (5.98) | 420.6 (4.53) | 8,360 | 02.78 x |
| nanoq | 11.93 (0.06) | 94.93 (0.45) | 36,775 | 12.22 x |
| nanoq-fast | 08.05 (0.05) | 03.90 (0.30) | 895,148 | 297.5 x |
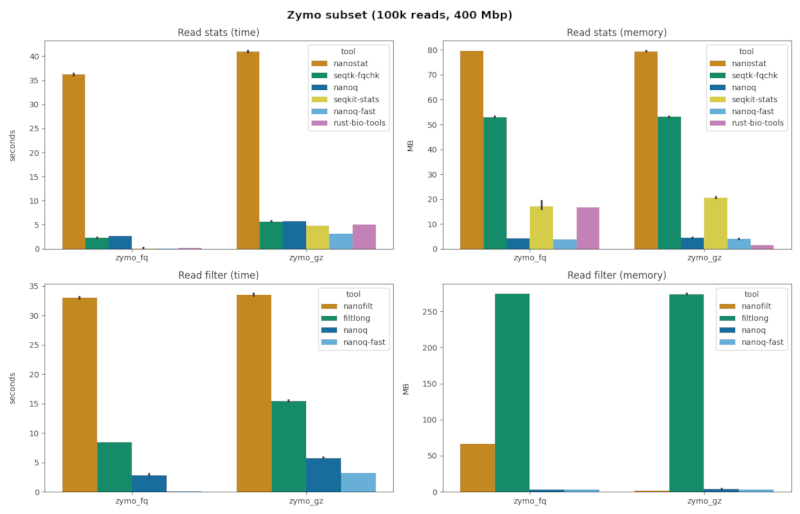
stats + zymo.fq
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 | 质量分数 |
|---|---|---|---|---|---|
| nanostat | 79.64 (0.14) | 36.22 (0.27) | 2,760 | 01.00 x | 真 |
| nanoq | 04.26 (0.09) | 02.69 (0.02) | 37,147 | 13.46 x | 真 |
| seqtk-fqchk | 53.01 (0.05) | 02.28 (0.06) | 43,859 | 15.89 x | 真 |
| seqkit-stats | 17.07 (3.03) | 00.13 (0.00) | 100,000 | 36.23 x | 假 |
| rust-bio | 16.61 (0.08) | 00.22 (0.00) | 100,000 | 36.23 x | 假 |
| nanoq-fast | 03.81 (0.05) | 00.08 (0.00) | 100,000 | 36.23 x | 假 |
stats + zymo.fq.gz
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 | 质量分数 |
|---|---|---|---|---|---|
| nanostat | 79.46 (0.22) | 40.98 (0.31) | 2,440 | 01.00 x | 真 |
| nanoq | 04.44 (0.09) | 05.74 (0.04) | 17,421 | 07.14 x | 真 |
| seqtk-fqchk | 53.11 (0.05) | 05.70 (0.08) | 17,543 | 07.18 x | 真 |
| rust-bio | 01.59 (0.06) | 05.06 (0.04) | 19,762 | 08.09 x | 假 |
| seqkit-stats | 20.54 (0.41) | 04.85 (0.02) | 20,619 | 08.45 x | 假 |
| nanoq-fast | 03.95 (0.07) | 03.15 (0.02) | 31,746 | 13.01 x | 假 |
filter + zymo.fq
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 |
|---|---|---|---|---|
| nanofilt | 66.29 (0.15) | 33.01 (0.24) | 3,029 | 01.00 x |
| filtlong | 274.5 (0.04) | 08.49 (0.01) | 11,778 | 03.89 x |
| nanoq | 03.61 (0.04) | 02.81 (0.28) | 35,587 | 11.75 x |
| nanoq-fast | 03.26 (0.06) | 00.12 (0.01) | 100,000 | 33.01 x |
filter + zymo.fq.gz
| 命令 | mb(标准差) | 秒(标准差) | 读段/秒 | 加速比 |
|---|---|---|---|---|
| nanofilt | 01.57 (0.07) | 33.48 (0.35) | 2,986 | 01.00 x |
| filtlong | 274.2 (0.04) | 16.45 (0.09) | 6,079 | 02.04 x |
| nanoq | 03.68 (0.06) | 05.77 (0.04) | 17,331 | 05.80 x |
| nanoq-fast | 03.45 (0.07) | 03.20 (0.02) | 31,250 | 10.47 x |
依赖项
Nanoq 使用 needletail 进行读操作,并使用 niffler 进行输出压缩。
词源
避免了与nanoqc的名称冲突,并将c删除,得到nanoq [nanɔq],巧合的是,这在北美(爱斯基摩-阿留申语,格陵兰语)中意为“北极熊”。如果您觉得nanoq对您的工作有帮助,请考虑向北极熊基金、RAVEN或因纽特人组织捐赠一小笔款项。
贡献
我们欢迎任何建议或pull请求。请随时在GitHub的仓库中提出问题。
依赖项
~7.5MB
~132K SLoC