2 个不稳定版本
| 0.2.0 | 2022年6月24日 |
|---|---|
| 0.1.1 | 2022年2月12日 |
#992 in 算法
1MB
1K SLoC
affinityprop
该 affinityprop crate 提供了亲和传播聚类算法的优化实现,该算法能够识别数据中的聚类,无需先验知识来了解数据中的聚类数量。原始算法由 Brendan Frey 和 Delbery Dueck 开发。
关于
亲和传播从数据集中识别一组代表性的示例,称为 原型。
简要来说,该算法接受一个描述所有数据值之间的 成对相似性 的矩阵作为输入。这些信息用于计算成对 责任 和 可用性。责任 r(i,j) 描述点 j 相对于其他潜在原型,作为点 i 的原型时有多适合。可用性 a(i,j) 描述点 i 接受点 j 作为其原型时相对于其他原型有多合适。
用户提供一系列 收敛迭代次数 以重复计算,之后从数据集中提取潜在原型。然后,算法继续重复,直到原型值停止变化,或达到 最大迭代次数。
为什么是这个 crate?
亲和传播的本质需要 O(n^2) 的时间复杂度。现有的 sklearn 版本使用 Python 库 numpy 实现,该库包含矢量化行操作。结合 SIMD 指令,这导致完成时间减少。
然而,在输入值较大的应用中,O(n2)的时间复杂度仍然具有约束性。本库使用rayon库实现了亲和力传播,这使得整体运行时间大幅降低——在发布模式下编译时,最多可降低30-60%!
依赖项
cargo,版本需满足rustc >=1.58
安装
在Rust代码中
[dependencies]
affinityprop = "0.2.0"
ndarray = "0.15.4"
作为命令行工具
cargo install affinityprop
用法
从Rust代码
affinityprop库期望一个类型,该类型定义了如何计算所有数据点的成对Similarity。本库提供了NegEuclidean、NegCosine和LogEuclidean结构体,分别定义为-1 * sum((a - b)**2)、-1 * (a . b)/(|a|*|b|)和sum(log((a - b)**2))。
建议希望以不同方式计算相似度的用户,亲和力传播期望s(i,j) > s(i, k)当且仅当i比j与k更相似时。
use ndarray::{arr1, arr2, Array2};
use affinityprop::{AffinityPropagation, NegCosine, Preference};
let x: Array2<f32> = arr2(&[[0., 1., 0.], [2., 3., 2.], [3., 2., 3.]]);
// Cluster using negative cosine similarity with a pre-defined preference
let ap = AffinityPropagation::default();
let (converged, results) = ap.predict(&x, NegCosine::default(), Preference::Value(-10.));
assert!(converged && results.len() == 1 && results.contains_key(&0));
// Cluster with list of preference values
let pref = arr1(&[0., -1., 0.]);
let (converged, results) = ap.predict(&x, NegCosine::default(), Preference::List(&pref));
assert!(converged);
assert!(results.len() == 2 && results.contains_key(&0) && results.contains_key(&2));
// Use damping=0.5, threads=2, convergence_iter=10, max_iterations=100,
// median similarity as preference
let ap = AffinityPropagation::new(0.5, 2, 10, 100);
let (converged, results) = ap.predict(&x, NegCosine::default(), Preference::Median);
assert!(converged);
assert!(results.len() == 2 && results.contains_key(&0) && results.contains_key(&2));
// Predict with pre-calculated similarity
let s: Array2<f32> = arr2(&[[0., -3., -12.], [-3., 0., -3.], [-12., -3., 0.]]);
let ap = AffinityPropagation::default();
let (converged, results) = ap.predict_precalculated(s, Preference::Value(-10.));
assert!(converged && results.len() == 1 && results.contains_key(&1));
从命令行
可以从命令行运行affinityprop,并用于分析数据文件
ID1 val1 val2
ID2 val3 val4
ID3 val5 val6
其中IDn是任何字符串标识符,valn是浮点数(十进制)值。文件分隔符由命令行中的-l标志提供。相似度将根据-s标志设置的选项进行计算。
对于没有行ID的文件
val1 val2
val3 val4
val5 val6
请从命令行提供-n标志。ID将自动通过零基索引分配。
用户还可以通过从命令行传递-s 3标志,并按以下方式组织其输入文件来提供预计算的相似度矩阵:
ID1 sim11 sim12 sim13
ID2 sim21 sim22 sim23
ID3 sim31 sim32 sim33
其中rowi、colj是输入i和j之间的成对相似度。
或者,对于没有行标签的文件,用户可以传递-n -s 3
sim11 sim12 sim13
sim21 sim22 sim23
sim31 sim32 sim33
ID将自动通过零基索引分配。
帮助菜单
affinityprop 0.2.0
Chris N. <christopher.neely1200@gmail.com>
Vectorized and Parallelized Affinity Propagation
USAGE:
affinityprop [OPTIONS] --input <INPUT>
FLAGS:
-n, --no_labels Input file does not contain IDS as the first column
-h, --help Prints help information
-V, --version Prints version information
OPTIONS:
-c, --convergence_iter <CONV_ITER> Convergence iterations, default=10
-d, --damping <DAMPING> Damping value in range (0, 1), default=0.9
-l, --delimiter <DELIMITER> File delimiter, default '\t'
-i, --input <INPUT> Path to input file
-m, --max_iter <MAX_ITER> Maximum iterations, default=100
-r, --precision <PRECISION> Set f32 or f64 precision, default=f32
-p, --preference <PREF> Preference to be own exemplar, default=median pairwise similarity
-s, --similarity <SIMILARITY> Set similarity calculation method
(0=NegEuclidean,1=NegCosine,2=LogEuclidean,3=precalculated), default=0
-t, --threads <THREADS> Number of worker threads, default=4
结果
结果以以下格式打印到stdout
Converged=true/false nClusters=NC nSamples=NS
>Cluster=n size=N exemplar=i
[comma-separated cluster member IDs/indices]
>Cluster=n size=N exemplar=i
[comma-separated cluster member IDs/indices]
...
运行时间和资源说明
亲和力传播在运行时间和内存方面都是O(n2)。本库旨在解决前者,而非后者。
给定以下条件,可以计算出估算的内存使用量
memory(GB) = p * 4 * N^2 / 2^30
对于 N 个输入。32位浮点精度时,p = 4,64位时,p = 8。
与 sklearn 实现的比较
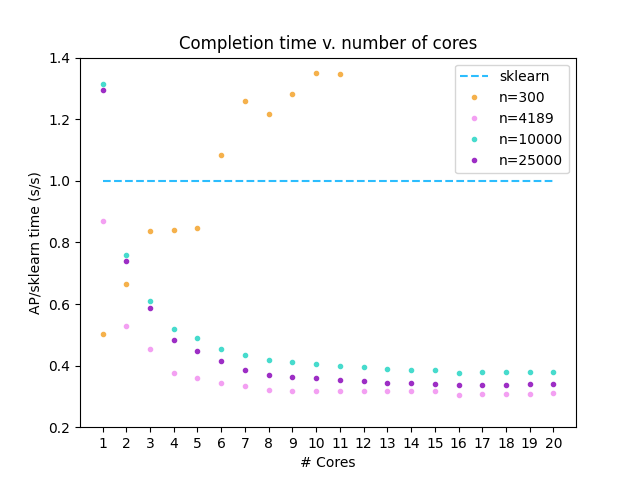
本实现已在 sklearn 的 make_blobs 函数生成的 50-D 各向同性高斯块(n=300,10000,25000)上进行了测试。还选择了一个具有生物学相关性的数据集(n=4189)(数据集)。与 make_blobs 提供的标签进行比较时,高斯数据的 ARI/F1 分数 >= 0.99。与基于 sklearn 的标签进行比较时,生物学数据集的 ARI/F1 = 0.98。
此 affinityprop 实现与 scikit-learn-1.0.2 中包含的 Affinity Propagation 实现进行了比较,并使用 numpy-1.22.2 和 Python 3.10.0 运行。此分析使用 Ryzen 9 3950X 处理器完成。
在所有分析中,阻尼系数=0.95,收敛迭代次数=400,最大迭代次数=4000。高斯数据偏好=-1000.0,生物数据偏好=-10.0。
依赖项
~3.5MB
~58K SLoC