9 个稳定版本
| 3.1.0 | 2024年7月16日 |
|---|---|
| 3.0.0 | 2023年6月17日 |
| 2.2.1 | 2023年1月9日 |
| 2.1.3 | 2022年10月4日 |
| 2.0.0 | 2022年5月3日 |
#164 在 数学
140KB
1K SLoC
🌲 PhyloDM
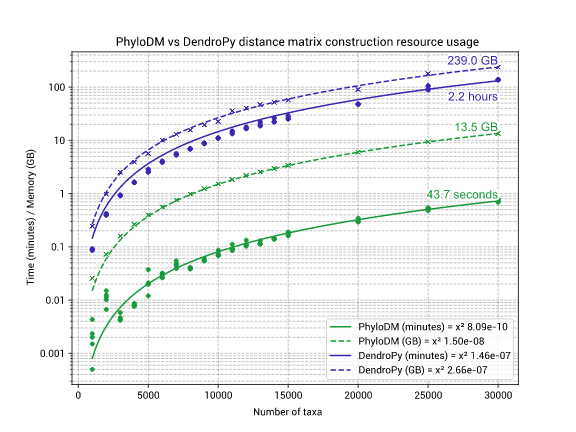
PhyloDM 是一个高性能库,可以将系统发育树转换为成对距离矩阵。
对于具有 30,000 个物种的树,PhyloDM 将使用
- ~14GB 的内存(比 DendroPy 低 94%)
- ~1 分钟的 CPU 时间(比 DendroPy 快 183 倍)。
PhyloDM 使用 Rust 编写,并通过 Python PyO3 API 公开。这意味着它可以在 Python 或 Rust 中使用,但下面的文档是为 Python 编写的。有关 Rust 文档,请参阅 Crates.io。
⚙ 安装
需要 Python 3.9+
PyPI
为大多数 64 位 Unix 平台打包了预编译的二进制文件。如果您在不同的平台上安装,则需要安装 Rust 来编译二进制文件。
python -m pip install phylodm
Conda
conda install -c b bioconda phylodm
🐍 快速入门
可以从 Newick 文件或 DendroPy 树创建成对距离矩阵。
from phylodm import PhyloDM
# PREPARATION: Create a test tree
with open('/tmp/newick.tree', 'w') as fh:
fh.write('(A:4,(B:3,C:4):1);')
# 1a. From a Newick file
pdm = PhyloDM.load_from_newick_path('/tmp/newick.tree')
# 1b. From a DendroPy tree
import dendropy
tree = dendropy.Tree.get_from_path('/tmp/newick.tree', schema='newick')
pdm = PhyloDM.load_from_dendropy(tree)
# 2. Calculate the PDM
dm = pdm.dm(norm=False)
labels = pdm.taxa()
"""
/------------[4]------------ A
+
| /---------[3]--------- B
\---[1]---+
\------------[4]------------- C
labels = ('A', 'B', 'C')
dm = [[0. 8. 9.]
[8. 0. 7.]
[9. 7. 0.]]
"""
访问数据
dm 方法生成一个对称的 NumPy 矩阵,并返回矩阵行/列顺序的键元组。
# Calculate the PDM
dm = pdm.dm(norm=False)
labels = pdm.taxa()
"""
/------------[4]------------ A
+
| /---------[3]--------- B
\---[1]---+
\------------[4]------------- C
labels = ('A', 'B', 'C')
dm = [[0. 8. 9.]
[8. 0. 7.]
[9. 7. 0.]]
"""
# e.g. The following commands (equivalent) get the distance between A and B
dm[0, 1] # 8
dm[labels.index('A'), labels.index('B')] # 8
归一化
如果 dm 的 norm 参数设置为 True,则数据将通过树中所有边的总和进行归一化。
⏱ 性能
测试是在 Intel(R) Xeon(R) CPU E5-2650 v3 @ 2.30GHz 上使用 scripts/performance/Snakefile 执行的。
对于大量物种,使用 PhyloDM 有益,但如果树中物种数量较少,则使用 DendroPy 更有益,因为它提供了大量功能。
依赖项
~6–12MB
~126K SLoC