5个版本 (3个重大更新)
使用旧的Rust 2015
| 0.4.0 | 2017年6月26日 |
|---|---|
| 0.3.0 | 2016年8月29日 |
| 0.2.0 | 2016年8月16日 |
| 0.1.1 | 2016年6月12日 |
| 0.1.0 | 2016年6月11日 |
1993在 算法
44KB
467 行
darwin-rs
这个库允许你使用Rust编程语言编写进化算法(EA)。
由Willi Kappler编写,许可证:MIT - 版本0.4 (2017.06.26)
文档: darwin-rs
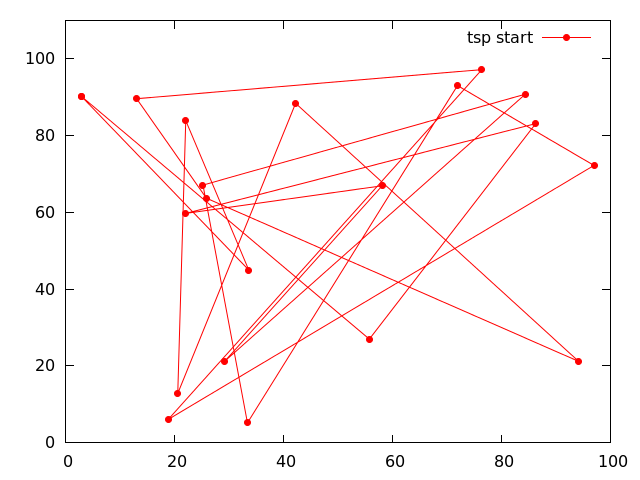
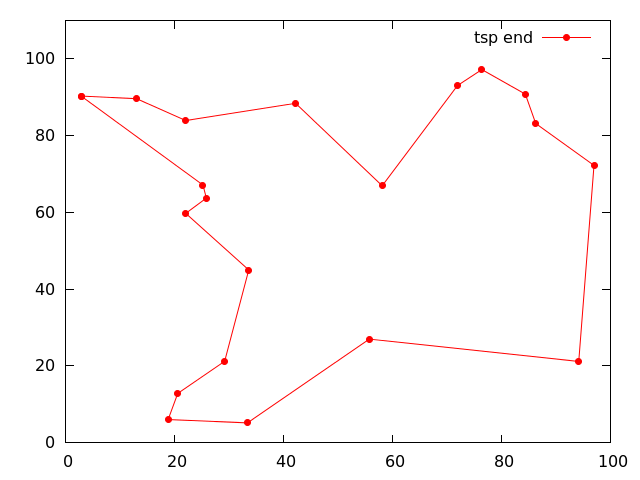
示例文件夹包含以下示例
- TSP(旅行商问题):进化算法的经典问题类型(见上面两张图片)。
- 数独:使用EA的数独求解器。
- 皇后:使用EA解决皇后问题。虽然不如这个快 ;-)
- OCR:一个简单的光学字符识别示例。在图像缓冲区上使用真型字体绘制(渲染)两个字符串,然后找到表示绘制的文本的完美匹配。
darwin-rs使用语义版本化
用法
将以下内容添加到您的项目中的Cargo.toml
[dependencies]
darwin-rs = "0.4"
并在您的应用程序的Rust源代码中添加以下内容
extern crate darwin_rs;
use darwin_rs::{Individual, SimulationBuilder, Population, PopulationBuilder, SimError};
基本上,您必须为您的数据结构实现Individual特质
#[derive(Debug, Clone)]
struct MyStruct {
text: String
}
impl Individual for MyStruct {
fn mutate(&mut self) {
// Mutate the struct here.
...
}
fn calculate_fitness(&mut self) -> f64 {
// Calculate how good the data values are compared to the perfect solution
...
}
fn reset(&mut self) {
// Resets all the data for this individual instance.
// This is done to avoid getting stuck in a local minimum.
...
}
}
需要这三个方法
mutate(&mut self):修改结构体的内容。
calculate_fitness(&mut self) -> f64:计算适应度值,即此个体结构实例与完美解的接近程度?数值越小表示越接近(==错误越少==离最优解越近)。
reset(&mut self):在特定数量的迭代后重置所有数据(见reset_limit),以避免局部最小值。
还有一个方法(new_fittest_found),但它是可选的,默认实现不执行任何操作。
如果您想在所有个体之间共享一个大的数据结构,您需要Arc,请参阅TSP和OCR示例。
现在您必须创建一个或多个具有不同属性的种群
// A helper function that creates a vector of individuals of your data structure:
let my_pop = make_population(100);
let population1 = PopulationBuilder::<MyStruct>::new()
.set_id(1)
.initial_population(&my_pop)
.increasing_exp_mutation_rate(1.03)
.reset_limit_increment(100)
.reset_limit_start(100)
.reset_limit_end(1000)
.finalize().unwrap();
let population2 = PopulationBuilder::<MyStruct>::new()
.set_id(2)
.initial_population(&my_pop)
.increasing_exp_mutation_rate(1.04)
.reset_limit_increment(200)
.reset_limit_start(100)
.reset_limit_end(2000)
.finalize().unwrap();
set_id():设置种群ID。这可以是任何正u32整数。目前这仅用于内部统计,例如:哪个种群拥有最健壮的个体?这可能有助于您设置正确的模拟参数。
initial_population():此方法接受一个个体向量。最佳实践是使用辅助函数,请参阅示例。
increasing_exp_mutation_rate(): 为每个个体设置突变率:使用指数突变率。
reset_limit_increment(): 每次迭代计数器达到限制时,增加此数量的重置限制。
reset_limit_start(): 重置限制的起始值。
reset_limit_end(): 重置限制的结束值。如果达到此结束值,则重置限制重置为上述起始值。
或者,您也可以将所有种群放入一个向量中。
之后,您需要创建一个新的模拟实例并设置参数。
let my_builder = SimulationBuilder::<MyStruct>::new()
.factor(0.34)
.threads(2)
.add_population(population1)
.add_population(population2)
.finalize();
match my_builder {
Err(SimError::EndIterationTooLow) => println!("more than 10 iteratons needed"),
Ok(mut my_simulation) => {
my_simulation.run();
println!("total run time: {} ms", my_simulation.total_time_in_ms);
println!("improvement factor: {}", my_simulation.simulation_result.improvement_factor);
println!("number of iterations: {}", my_simulation.simulation_result.iteration_counter);
my_simulation.print_fitness();
}
}
factor(): 设置终止条件:如果改进因子比这个值好或等于这个值,则模拟停止。
threads(): 用于模拟的线程数。
add_population(): 这会将之前创建的种群添加到模拟中。
finalize(): 完成设置并进行合理性检查。如果没有配置错误,则返回 Ok(Simulation)。
add_muliple_populations(): 允许您在一个方法调用中添加向量中的所有种群。
然后,只需对 finalize() 的结果进行匹配,并调用 simulation.run() 以启动模拟。完成后,您可以访问一些统计信息(total_time_in_ms、improvement_factor、iteration_counter)以及当然种群。
for population in my_simulation.habitat {
for wrapper in population.population {...}
}
每个个体都封装在一个包含模拟所需额外信息的 Wrapper 结构体中:适应度 和 突变次数。有关完整的工作程序示例,请参阅示例文件夹。
讨论
使用的 crate
- jobsteal: 并行化
- error-chain: 简单的错误处理
- log: 使用日志机制而不是
println!()
类似的 crate
- genetic-files
- RsGenetic
- evo-rs
- calco-rs
- GenGen
- parasailors
- random-wheel-rs
- roulette-wheel-rs
- differential-evolution-rs
- evolve-sbrain
- Nodevo
任何反馈都受欢迎!
依赖关系
~3–4.5MB
~84K SLoC