6 个版本
| 0.2.4 | 2024年4月24日 |
|---|---|
| 0.2.3 | 2021年4月9日 |
| 0.2.2 | 2021年3月24日 |
| 0.1.0 | 2021年3月3日 |
#762 在 解析器实现
324 每月下载量
90KB
1.5K SLoC
neuroformats
处理 Rust 的结构神经影像文件格式。
通过实现各种文件格式的解析器,neuroformats 包提供在 Rust 中访问结构神经影像数据的功能。重点是表面基于的脑形态计量数据,如神经影像软件套件(如 FreeSurfer,CAT12 等)从 3D 或 4D 磁共振成像 (MRI) 数据生成的数据。
背景
在基于表面的神经影像中,基本上是从分割的 3D 脑图像重建代表人类皮层 3D 结构的网格。然后,从重建中计算人类大脑的特性,如特定位置的皮层厚度,并将其作为网格的顶点数据存储。
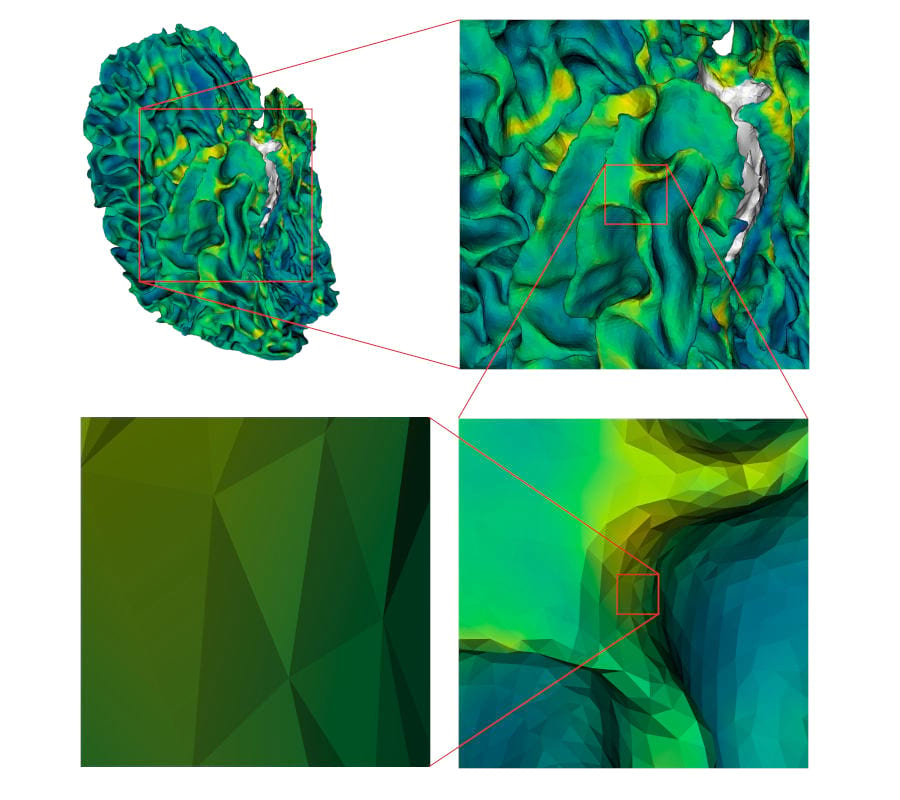
图 1 一个代表人类大脑白质表面的网格,使用 viridis 色彩映射将其上的皮层厚度值映射。显示不同的放大倍数,最后一张图中可以识别三角形和顶点位置。
安装
neuroformats 包在 crates.io 上。
要将库用于您的项目,请将其作为依赖项添加到您的 Cargo.toml 文件中,例如:
[dependencies]
neuroformats = "0.2.3"
功能
- 读取和写入 FreeSurfer 每个顶点数据在 curv 格式(如
subject/surf/lh.thickness):函数neuroformats::read_curv和write_curv - 读取和写入大脑网格在 FreeSurfer 二进制网格格式(如
subject/surf/lh.white):read_surf和write_surf - 读取和写入 FreeSurfer 标签文件(如
subject/label/lh.cortex.label):read_label和write_label - 读取FreeSurfer脑表面分区(如
subject/label/lh.aparc.annot):read_annot - 读取和写入FreeSurfer脑体积和其他来自MGH和MGZ文件的数据:
read_mgh和write_mgh
实现了各种实用函数,用于在返回的结构体上执行常见计算,例如从MGH头部数据计算vox2ras矩阵或在脑表面分区中查找属于特定脑图谱区域的全部顶点。
文档
API文档
可以在 docs.rs/neuroformats 找到 neuroformats API文档。
简短使用示例
读取由FreeSurfer计算的顶点级皮质厚度
use neuroformats::read_curv;
let curv = read_curv("subjects_dir/subject1/surf/lh.thickness").unwrap();
let thickness_at_vertex_0 : f32 = curv.data[0];
现在你有一个包含皮质厚度值的 Vec<f32>,存储在 curv.data 中。值的顺序与相应脑表面重建的顶点顺序相匹配(例如,在 subjects_dir/subject1/surf/lh.white 中的左脑半球白色表面网格)。
完整演示应用程序
有一个小型命令行演示应用程序,它从脑表面网格中加载图像,并基于网格将图像射影到PNG文件。该演示应用程序位于 ./examples/brain_rpt 目录。该演示使用 Eric Zhang和Alexander Morozov的rpt crate 进行射影。
有关更多示例,请参阅 neuroformats API文档 和 源代码中的单元测试。
开发信息
请参阅 README_DEV文件。
许可证
neuroformats crate是免费软件,根据MIT或APACHE-2许可证双授权。
贡献
欢迎贡献。在进行重大更改之前请先联系,以避免浪费精力。
帮助和联系
如果您想讨论某些内容、需要帮助或发现错误,请在此GitHub上打开一个问题。
neuroformats crate是由 Tim Schäfer 编写的。如果您需要与我联系,可以在我的网站上找到我的电子邮件地址。
依赖关系
~5.5MB
~101K SLoC